J'utilise un kit d'extraction (ARN en l'occurrence, puis je les reverse-transcris), on lyse juste les tissus au sonicateur (ou à l'azote mais j'aime moins). J'amplifie mon bout de COI avec ces primers, purification. Je refais une PCR avec les mêmes amorces pour préparer le Sanger et j'envoie à la plate-forme de séquençage.
Lors de votre inscription suivez impérativement ces recommandations pour que votre compte soit validé.
Si vous voyez ce bandeau, c'est que vous n'êtes pas connecté. En cas de difficultés de connexion cliquez d’abord sur CE LIEN avant de cliquer sur “Connexion”
Si vous voyez ce bandeau, c'est que vous n'êtes pas connecté. En cas de difficultés de connexion cliquez d’abord sur CE LIEN avant de cliquer sur “Connexion”
[Jaera hopeana] Inconnu clandestin
Animateur : noelouigre1
- DrPorcellion
- Membre
- Enregistré le : vendredi 18 septembre 2015, 12:57
- Localisation : Nantes
[Jaera hopeana] Inconnu clandestin
Si, si, du Sanger 
J'utilise un kit d'extraction (ARN en l'occurrence, puis je les reverse-transcris), on lyse juste les tissus au sonicateur (ou à l'azote mais j'aime moins). J'amplifie mon bout de COI avec ces primers, purification. Je refais une PCR avec les mêmes amorces pour préparer le Sanger et j'envoie à la plate-forme de séquençage.
J'utilise un kit d'extraction (ARN en l'occurrence, puis je les reverse-transcris), on lyse juste les tissus au sonicateur (ou à l'azote mais j'aime moins). J'amplifie mon bout de COI avec ces primers, purification. Je refais une PCR avec les mêmes amorces pour préparer le Sanger et j'envoie à la plate-forme de séquençage.
Balises :
- Mazancourt
- Animateur
- Enregistré le : vendredi 6 novembre 2015, 13:26
- Localisation : Paris
[Jaera hopeana] Inconnu clandestin
Je séquence aussi mes bêtes en Sanger au Muséum 
Pour le BLAST c'est peut-être une contamination, tu as envoyé des produits PCR d'autres bêtes à séquencer en même temps ?
Ou sinon, ce sont les séquences déposées sur GenBank qui ont été mal identifiées, après tout ça s'est déjà vu, et comme il n'y a aucun contrôle...
Pour le BLAST c'est peut-être une contamination, tu as envoyé des produits PCR d'autres bêtes à séquencer en même temps ?
Ou sinon, ce sont les séquences déposées sur GenBank qui ont été mal identifiées, après tout ça s'est déjà vu, et comme il n'y a aucun contrôle...
Valentin.
- DrPorcellion
- Membre
- Enregistré le : vendredi 18 septembre 2015, 12:57
- Localisation : Nantes
[Jaera hopeana] Inconnu clandestin
Je ne pense pas que ça soit de la contamination ... mais je n'ai pas de preuve 
Les erreurs d'identification genbank sont possibles aussi mais là c'est tellement gros (isopode marin/terrestre) que ce n'est pas non plus la piste que je privilégie.
J'ai un collègue qui a eu le même type de problème avec son blast... je garde mes soupçons sur la séquence en elle même (insert mito dans le noyau ?! pas la même portion dugène ?) mais je n'ai pas le temps de creuser l'histoire pour le moment. Si j'ai du nouveau je vous dirai bien-sûr.
Les erreurs d'identification genbank sont possibles aussi mais là c'est tellement gros (isopode marin/terrestre) que ce n'est pas non plus la piste que je privilégie.
J'ai un collègue qui a eu le même type de problème avec son blast... je garde mes soupçons sur la séquence en elle même (insert mito dans le noyau ?! pas la même portion du
- entonantes44
- Membre confirmé
- Enregistré le : dimanche 20 juillet 2014, 21:10
- Localisation : Gironde / Charente Maritime
[Jaera hopeana] Inconnu clandestin
Cela m'étonne un peu aussi. Je n'ai sans doute pas assez de recul pour bien analyser. Par contre, ce qui pourrait se faire quand tu auras du temps c'est de reconstruite un arbre à partir de tes séquences et voir si cela peut être cohérent. Cela te donnera une idée de la qualité de tes séquences.
C'est intéressant en tout cas, merci de nous avoir proposer ces premières séquences . C'est quoi le nom de vos plateformes de séquençages? cela doit quand même être du Sanger nouvelle génération à mon avis, pas la méthode originelle.
. C'est quoi le nom de vos plateformes de séquençages? cela doit quand même être du Sanger nouvelle génération à mon avis, pas la méthode originelle.
C'est intéressant en tout cas, merci de nous avoir proposer ces premières séquences
Brendan
- Mazancourt
- Animateur
- Enregistré le : vendredi 6 novembre 2015, 13:26
- Localisation : Paris
[Jaera hopeana] Inconnu clandestin
Chez nous, on passe par l'entreprise Eurofins 
Valentin.
- entonantes44
- Membre confirmé
- Enregistré le : dimanche 20 juillet 2014, 21:10
- Localisation : Gironde / Charente Maritime
[Jaera hopeana] Inconnu clandestin
D'accord, oui moi aussi je passais par eux. C'est donc bien un technique encore en cours, couplée à l'électrophorèse capillaire. Sanger : deux prix Nobel, c'est correct 
Brendan
- guillaume
- Animateur—Admin-galerie
- Enregistré le : dimanche 7 mars 2004, 13:00
- Localisation : Annemasse (Haute-Savoie)
[Jaera hopeana] Inconnu clandestin
Petite question bête: Jaera hopeana est-il bien dans la base de donnée des organismes séquencés (mitochondrial) pour le blast? Je ne trouve pas la sequence de la cox1 sur ncbi...
- entonantes44
- Membre confirmé
- Enregistré le : dimanche 20 juillet 2014, 21:10
- Localisation : Gironde / Charente Maritime
[Jaera hopeana] Inconnu clandestin
Non, il n'y est pas. Ou alors ce serait une surprise. Mais par contre il y a quelques isopodes et Benjamin aurait bien voulu avoir un joli petit arbre propre sur lequel positionner sa séquence. Mais là c'est un peu étonnant, cela sort pour un Amphipode alors qu'il y a de l'isopode et avec un résultat étonnant!
Brendan
- guillaume
- Animateur—Admin-galerie
- Enregistré le : dimanche 7 mars 2004, 13:00
- Localisation : Annemasse (Haute-Savoie)
[Jaera hopeana] Inconnu clandestin
C'est effectivement très étonnant 
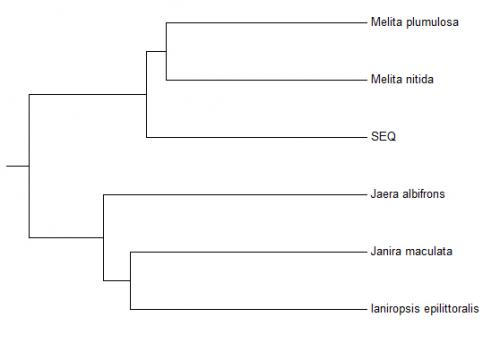
Par curiosité j'ai fait un alignement de différentes espèces de Janiridae et de Melitidae, en excluant Melita palmata (au cas où l'erreur vienne de Genebank), puis une reconstruction de laphylogénie avec le logiciel Mega7.
Voilà le résultat (SEQ = séquence de notre spécimen):
Cela confirme que le problème ne vient pas de Genbank...
Guillaume JACQUEMIN : France : Les Portes-en-Ré : 17880 : 09/10/2015
Altitude : NR - Taille : 1 mm
Réf. : 176948
Par curiosité j'ai fait un alignement de différentes espèces de Janiridae et de Melitidae, en excluant Melita palmata (au cas où l'erreur vienne de Genebank), puis une reconstruction de la
Voilà le résultat (SEQ = séquence de notre spécimen):
Cela confirme que le problème ne vient pas de Genbank...

Guillaume JACQUEMIN : France : Les Portes-en-Ré : 17880 : 09/10/2015
Altitude : NR - Taille : 1 mm
Réf. : 176948
- entonantes44
- Membre confirmé
- Enregistré le : dimanche 20 juillet 2014, 21:10
- Localisation : Gironde / Charente Maritime
[Jaera hopeana] Inconnu clandestin
C'est malin, c'est propre, j'aime  .
.
Je n'avais pas trouvé toutes ces séquences je dois t'avouer.
Cela précise notre problème. Qu'en dis-tu Benjamin?
Je n'avais pas trouvé toutes ces séquences je dois t'avouer.
Cela précise notre problème. Qu'en dis-tu Benjamin?
Brendan
